Dell EMC Ready oplossing voor HPC-biowetenschappen: BWA-GATK Pijplijndoorvoertests met Cascade Lake CPU en Lustre ME4 Refresh
Summary: Archivering als artikel is gebaseerd op HPC-documentatie die niet langer online wordt gehost, en het artikel kan niet worden bewerkt om aan de verwachtingen van de KB te voldoen Dell EMC Ready oplossing voor HPC-biowetenschappen: BWA-GATK Pijplijndoorvoertests met Cascade Lake CPU en Lustre ME4 Refresh ...
Symptoms
Knooppuntconfiguratie met 64 rekeneenheden van Dell EMC Ready Solutions voor HPC-biowetenschappen kan 194 genomen per dag verwerken (50x dekkingsdiepte).
Overzicht
Variant aanroepen
Het doel van deze blog is om waardevolle prestatie-informatie te geven over de Intel® Xeon® Gold 6248 processor voor de BWA-GATK-pipelinebenchmark met Dell EMC Ready Solutions voor HPC Lustre Storage (ME4 serie refresh) (2). De Xeon® Gold 6248 CPU beschikt over 20 fysieke cores of 40 logische cores bij gebruik van hyper threading. De configuraties van de testclusters zijn samengevat in Tabel 1.
| Dell EMC PowerEdge C6420 | |
|---|---|
| CPU | 2 x Xeon® Gold 6248, 20 cores, 2,5 GHz (Cascade Lake) |
| RAM | 12 x 16 GB bij 2933 MTps |
| BESTURINGSSYSTEEM | RHEL 7.6 |
| Interconnect | Intel® Omni-Path |
| BIOS-systeemprofiel | Prestaties geoptimaliseerd |
| Logische processor | Disabled |
| Virtualisatietechnologie | Disabled |
| BWA | 0.7.15-R1140 |
| Samtools | 1.6 |
| GATK | 3,6-0-g89b7209 |
De geteste rekenknooppunten zijn via Intel® Omni-Path verbonden met Dell EMC Ready Solutions voor HPC Lustre Storage. De overzichtsconfiguratie van de storage wordt vermeld in Tabel 2.
Tabel 2 Hardware- en softwarespecificaties van de oplossing
| Dell EMC Ready Solution voor Lustre Storage | |
|---|---|
| Aantal knooppunten | 1 x Dell EMC PowerEdge R640 als Integrated Manager for Lustre (IML) 2 x Dell EMC PowerEdge R740 als metadataserver (MDS) 2 x Dell EMC PowerEdge R740 als objectstorageserver (OSS) |
| Processoren | IML-server: Dubbele Intel Xeon Gold 5118 @ 2,3 GHz MDS- en OSS-servers: Dual Intel Xeon Gold 6136 @ 3,00 GHz |
| Geheugen | IML-server: 12 x 8 GB, 2666 MT/s DDR4 RDIMM's, MDS- en OSS-servers: 24 x 16 GiB 2666 MT/s DDR4 RDIMM's |
| Externe storagecontrollers |
2 x Dell 12 Gb/s SAS HBA's (op elke MDS) 4 x Dell 12 Gb/s SAS HBA's (op elke OSS) |
| Objectstoragebehuizingen |
4 x ME4084 met een totaal van 336 x 8 TB NL 7,2K rpm SAS HDD's |
| Opslagbehuizing voor metadata |
1 x ME4024 met 24 x 960 GB SAS SSD's. Ondersteunt tot 4,68 B inodes |
| RAID-controllers | Duplex SAS RAID-controllers in de ME4084- en ME4024-behuizingen |
| Besturingssysteem | CentOS 7.5 x86_64 Red Hat Enterprise Linux (RHEL) 7.5 x86_64 |
| BIOS-versie | 1.4.5 |
| Intel Omni-Path IFS-versie |
10.8.0.0 |
| Versie van het Lustre-bestandssysteem |
2.10.4 |
| IML-versie | 4.0.7.0 |
De testgegevens werden gekozen uit een van Illumina's platina-genomen. ERR194161 is verwerkt met Illumina HiSeq 2000 ingediend door Illumina en kan worden verkregen bij EMBL-EBI. De DNA-identificatie voor dit individu is NA12878. Uit de beschrijving van de gegevens van de gelinkte website blijkt dat dit monster een >dekkingsdiepte van 30x heeft.
Prestatie-evaluatie
Prestaties van één voorbeeld van meerdere knooppunten
In afbeelding 1 wordt de runtime in een groot aantal samples en compute nodes met 50x Whole Genome Sequencing (WGS)-data samengevat. De tests die hier worden uitgevoerd, zijn ontworpen om prestaties op serverniveau aan te tonen, niet voor vergelijkingen op afzonderlijke componenten. De datapunten in figuur 1 zijn berekend op basis van het totale aantal samples, één sample per compute node (X-as in de afbeelding) die gelijktijdig worden verwerkt. De details van de informatie over de BWA-GATEK-pijpleiding zijn te vinden op de website van het Broad Institute (3). Het maximale aantal rekenknooppunten dat voor de tests wordt gebruikt, is 64x C6420's. C6420s met Lustre ME4 vertonen een beter schaalgedrag dan Lustre MD3.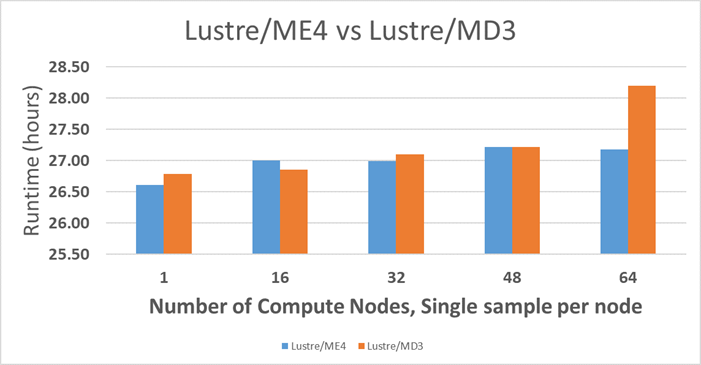
Afbeelding 1 Prestatievergelijkingen tussen Lustre MD3 en Lustre ME4
Prestaties van meerdere voorbeelden en meerdere knooppunten
Een typische manier om de NGS-pipeline uit te voeren, is door meerdere voorbeelden uit te voeren op een rekenknooppunt en meerdere rekenknooppunten te gebruiken om de doorvoer van het NGS-gegevensproces te maximaliseren. Het aantal rekenknooppunten dat voor de tests wordt gebruikt, is 64 van de C6420-rekenknooppunten en het aantal voorbeelden per knooppunt is vijf voorbeelden. Tot 320 monsters worden tegelijkertijd verwerkt om het maximale aantal genomen per dag te schatten zonder dat een taak mislukt.Zoals weergegeven in afbeelding 2, kan één C6420-rekenknooppunt 3,24 van de 50x volledige menselijke genomen per dag verwerken wanneer 5 monsters tegelijkertijd worden verwerkt. Voor elk monster worden 7 cores en 30 GB geheugen toegewezen.
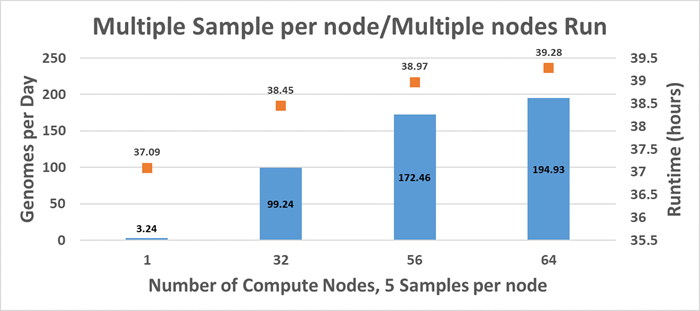
Afbeelding 2 Doorvoertests met maximaal 64 C6420's en de Lustre ME4
320 van de 50x volledige menselijke genomen kunnen in 40 uur worden verwerkt met 64 van de C6420 compute-knooppunten. Met andere woorden, de prestaties van de testconfiguratie komen neer op 194 genomen per dag voor het hele menselijke genoom met een dekkingsgraad van 50x.
Conclusie
Omdat de gegevensgrootte van WGS voortdurend is gegroeid. De huidige gemiddelde grootte van WGS is 50x. Dit is 5 keer groter dan een typische WGS 4 jaar geleden, toen we begonnen met het benchmarken van de BWA-GATT-pijplijn. De toename van het aantal data leidt niet tot extra druk op de capaciteit aan de storagezijde, omdat de meeste applicaties in de pipeline ook worden begrensd door de CPU-kloksnelheid. Daarom blijft de pipeline met een groeiende dataomvang langer lopen in plaats van dat er meer schrijfbewerkingen worden gegenereerd.Er wordt echter een groter aantal tijdelijke bestanden gegenereerd tijdens het proces omdat er meer gegevens moeten worden geparallelliseerd, en dit grotere aantal tijdelijke bestanden dat tegelijkertijd wordt geopend, put de limiet voor open bestanden in een Linux-besturingssysteem uit. Een van de applicaties wordt stilletjes niet voltooid door de limiet van het aantal geopende bestanden te bereiken. Een eenvoudige oplossing is om de limiet te verhogen naar >150K.
Desalniettemin heeft de Ready Solution met Lustre ME4 als scratch space een betere doorvoercapaciteit dan de vorige versie. Ready Solution met 64 knooppunten markeert 194 genomen per dag verwerkingskracht voor 50x WGS.
Bronnen
1. Een overzicht van tools voor variantanalyse van genoomsequentiegegevens van de volgende generatie. Pabinger S, Dander A, Fischer M, Snajder R, Sperk M, Efremova M, Krabichler B, Speicher MR, Zschocke J, Trajanoski Z. 2, s.l. : Korte Bioinform, 2014 maart, Vol. 15 (2). 10.1093/bib/bbs086.2. Dell EMC Ready oplossing voor HPC Glansre Storage. (Artikel niet langer beschikbaar ter referentie, opgehaald door HPC-team)
3. Toolkit voor genoomanalyse. https://software.broadinstitute.org/gatk/

Cause
Archivering als artikel is gebaseerd op HPC-documentatie die niet langer online wordt gehost, en het artikel kan niet worden bewerkt om aan de verwachtingen van de KB te voldoen
Resolution
Archivering als artikel is gebaseerd op HPC-documentatie die niet langer online wordt gehost, en het artikel kan niet worden bewerkt om aan de verwachtingen van de KB te voldoen