PowerEdge: Akselerering av genomisk dataanalyse med NVIDIA Clara Parabricks med Dell EMC DSS 8440-serveren og NVIDIA T4 GPU-er
Summary: Denne artikkelen inneholder informasjon om hvordan du akselererer genomisk dataanalyse ved hjelp av NVIDIA Parabricks på Dell EMC DSS 8440 med NVIDIA T4 GPU-er.
Instructions
Oversikt
Det første trinnet for behandling av Next Generation Sequencing-data (NGS) kalles primæranalyse. Dette trinnet gjelder for sekvensinstrumentet og genererer flere FASTQ-filer som inneholder sekvenslesinger. I det neste trinnet, kjent som sekundæranalyse, vil FASTQ-sekvensene leses inn i et referansegenom eller et referansetranskriptom. Mer behandling identifiserer varianter, eller forskjeller, mellom utvalget av interesse og en referanse. Variantene er kommentert og tolkes i påfølgende trinn. Det sekundære analysetidspunktet for ett enkelt eksempel strekker seg fra timer til dager, avhengig av datastørrelse, tilgjengelige dataressurser, programvare og analytisk arbeidsflyt.
Sekundæranalyse er en databehandlings- og lagringsintensiv prosess, særlig ved behandling av hundrevis til tusenvis av genomer. Det finnes mange strategier for å unngå flaskehalsene for sekundæranalyse. Inntil nylig har benyttelsen av maskinvareakselerasjon som bruker GPU-er eller FPGA-er forblitt lav på grunn av tilpasset programvare. Parabricks'genomikkprogramvare, som ble kjøpt opp av NVIDIA i 2019, har vært banebrytende for en programvarestabel som utfører forskjellige genomiske analysearbeidsflyter med GPUer. Vi testet Parabricksfor omtrent to år siden. Dell introduserte mange teknologiske fremskritt i sine servere og lagringsløsninger, og NVIDIA Clara Parabricks har gitt ut robuste versjoner med forbedret akselerasjon og tillegg av variantinnringere. For eksempel så en multi-GPU-serverdesign basert på Dell EMC DSS 8440-serveren med NVIDIA® Tesla® T4 GPU-er lovende ut for å akselerere sekundær analyse samtidig som den ga en attraktiv balanse mellom pris og ytelse. Denne bloggen rapporterer en ny referansearkitektur og resultater for referansemålinger for NVIDIA Clara Parabricks sekundær analyse på en multi-Tesla® T4 GPU, DSS 8440-server med Dell Isilon F800-lagring
Referansearkitektur
Figuren 1 illustrerer den testede referansearkitekturen. Arkitekturen er modulær og enkel å skalere. The NVIDIA Clara Parabricks applikasjonsprogramvare bruker en eller flere GPUer som gjør utskalering så enkelt som mulig. Maskinvarebyggeblokkene består av Dell PowerEdge R640 som administrasjonsnode, DSS 8440-server for GPU-databehandling og Dell EMC Isilon F800-lagring. 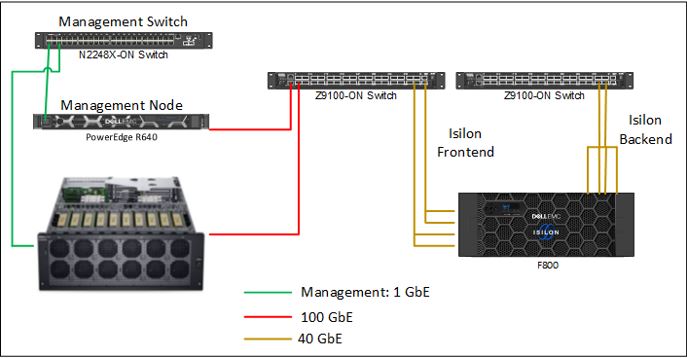
Figur 1Referansearkitektur blir testet
DSS 8440, 2 kontakter, 4U-server kan ta opptil 10 bransjeledende NVIDIA® Tesla® V100S Tensor-kjerne GPU-er, opptil 10 NVIDIA® Quadro RTX™ GPU-er eller opptil 16 NVIDIA Tesla T4 GPU-er, som leverer enorme hestekrefter. Den detaljerte konfigurasjonen av DSS 8440 er oppført i tabell 1.
| Dell EMC DSS 8440 | |
|---|---|
CPU |
2x Xeon® Gold 6248R 24 cores 3.0 GHz |
RAM |
24x 64GB at 2933 MTps |
Operating System |
Red Hat Enterprise Linux Server release 7.4 (Maipo) |
BIOS System Profile |
Performance Optimized |
Logical Processor |
Disabled |
Virtualization Technology |
Disabled |
Accelerators |
16x NVIDIA® Tesla® T4 GPUs |
Parabricks |
v3.0.0.05 |
To Z9100-switcher leverte sammenkoblingen mellom compute-noden og Isilon F800 Storage-klyngen. En ekstra bryter, N2248X-ON, brukes for administrasjon.
NGS-data
Data for benchmarking av sekundær analysekjøretid besto av tre Human, whole-genome sequencing (WGS) datasett, ERR091571


Ytelsesevaluering
Programvareforbedringer reduserer kjøretiden.
NVIDIA fortsetter å introdusere programvareforbedringer for NVIDIA Clara Parabricks. Figur 2 viser kjøretidsreduksjonen mellom to versjoner av Parabricks kjøre germline-pipelinen ved hjelp av Dell PowerEdge C4140-serveren med 4x V100 GPUs testmiljø. Overgangen fra v2.1.0 til v3.0.0 reduserte kjøretiden med 42 %.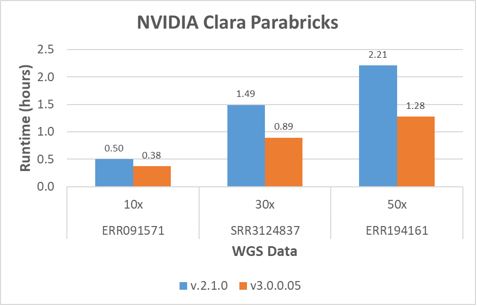
Figur 2: Siste versjon av Parabricks germline-varianten som kaller pipeline-kjøretid.
Ytelse for DSS 8440 med 16x T4s
Kjøretiden for en NVIDIA Clara Parabricks sekundæranalyse ved bruk av en enkelt T4 GPU er omtrent 30 % tregere enn ved bruk av en V100 GPU. To (2) T4 GPU-er gir imidlertid omtrent 10 % flere TFLOPS enn én (1) V100 GPU med omtrent halvparten av kostnadene. DSS 8440 har opptil 16 PCIe-spor, noe som åpner muligheten for å designe en T4 GPU-basert server som leverer lignende kjøretidsytelse som et C4140-system med fire V100 GPU-er, men til en lavere kostnad.
Den Parabricks Germline-analysen ble utført med en PowerEdge DSS 8440 med 16 T4-GPU-er. For hvert WGS-eksempeldatasett som ble beskrevet tidligere, ble kjøretiden registrert ved hjelp av 1, 2, 4, 8 og 16 T4 GPU-er per sekundær analyse. Resultatene tegnes inn i figurene 3 til 5. Generelt skal kjøretiden ikke skaleres lineært etterhvert som antallet GPU-er for hver analyse øker. Skaleringsmønsteret ligner på mengden av data per samplingsøkning fra 10x- til 50x-dekning.
Selv om det ikke er presentert her, ble en tidligere Dell EMC-undersøkelse av Parabricks Kjøretidsresultater med åtte eller flere V100 GPU-er per analyse skalerte ikke like effektivt som T4 GPU-ene. Flere tester viste at 6 T4 GPU-er genererte kjøretidsresultater som var nesten identiske med 4 V100 GPU-er.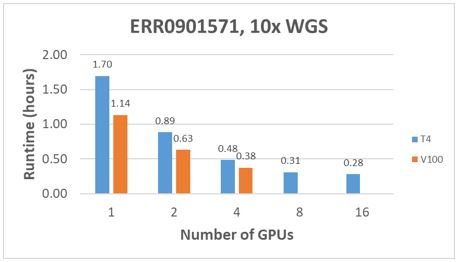
Figur 3 – Ytelsesammenligninger med 10 x WGS
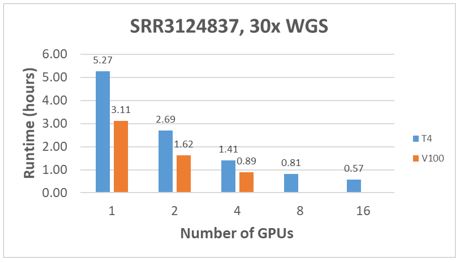
Figur 4 – Ytelsesammenligninger med 30 x WGS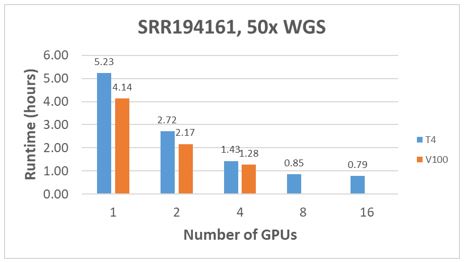
Figur 5 – Ytelsessammenligninger med 50x WGS
Konklusjon
En DSS 8440 med seksten T4 GPUer kan behandle tretti 50x menneskelige genomer per dag. En lignende daglig analyse av gjennomstrømming ved hjelp av en tradisjonell x86 CPU-arkitektur krever ti PowerEdge C6420e Compute-noder. Den fullstendige arkitekturen diskuteres i Dell Ready Solution for HPC Life Sciences: BWA-GATK Pipeline-gjennomstrømmingstester med Cascade Lake CPU og Lustre ME4 Refresh.
Dedikasjon av alle 16 T4 GPU-er for å behandle ett utvalg gir imidlertid få fordeler, siden bruk av 16 GPU-er per analyse i beste fall er 10 % raskere enn bruk av 8 GPU-er. Utformingen av DSS 8440 tillater flere sekundære analyser parallelt. Ved å tildele åtte T4 GPU-er for hver prøve øker den daglige analysegjennomstrømningen til ~50 genomer per dag. Bruk av fire GPU-er for hvert av eksemplene øker analysegjennomstrømmingen til ~70 genomer per dag. Viktigere er denne daglige produksjonen ved hjelp av T4 GPU-er mindre enn halve kostnaden ved bruk av V100 GPU-design.
I tillegg til hastigheten, er kompatibilitet med andre analyseverktøy viktig for sammenlignbare resultater. Informasjonen i Parabricks germline analyseresultater er nesten identiske med den velkjente BWA-GATK Haplotype innringeranalysen fra tidligere testing. Vi ønsket også å sammenligne Parabricks-variantens kalleresultater med andre verktøysett som samtools/mpileup. Disse to forskjellige verktøyene når ~ 90% samlet enighet for identifiserte varianter, og variasjoner i mange kjente genomiske regioner som inneholder viktige gener er mer enn 99%.