PowerEdge: przyspieszanie analizy danych genomu za pomocą oprogramowania NVIDIA Clara Parabricks, serwera Dell EMC DSS 8440 i procesorów graficznych NVIDIA T4
Summary: Ten artykuł zawiera informacje na temat przyspieszania analizy danych genomowych przy użyciu oprogramowania NVIDIA Parabricks na Dell EMC DSS 8440 z kartami graficznymi NVIDIA T4.
Instructions
Omówienie
Pierwszy krok przetwarzania danych sekwencjonowania nowej generacji (Next Generation Sequencing, NGS) nazywa się analizą podstawową. Ten krok jest specyficzny dla przyrządu do sekwencjonowania i generuje wiele plików FASTQ zawierających dane odczytów sekwencjonowania. W następnym kroku, zwanym analizą wtórną, dane odczytów sekwencjonowania FASTQ są mapowane na genom referencyjny lub transkryptom porównawczy. Dalsze przetwarzanie identyfikuje warianty lub różnice między próbką będącą przedmiotem zainteresowania a referencją. Poszczególne warianty są opatrzone adnotacjami i interpretowane w kolejnych etapach. Czas trwania analizy wtórnej pojedynczej próbki mieści się w zakresie od godzin do dni w zależności od rozmiaru danych, dostępnych zasobów komputerowych, oprogramowania i przepływu pracy analizy.
Analiza wtórna to proces wymagający użycia dużej mocy obliczeniowej i pamięci, szczególnie w przypadku przetwarzania setek czy tysięcy genomów. Istnieje wiele strategii zapobiegania problemów z wydajnością analizy wtórnej. Do niedawna korzystanie z akceleracji sprzętowej przy użyciu procesora GPU lub FPGA pozostawało na niskim poziomie ze względu na niestandardowe oprogramowanie wymagane przez akceleratory sprzętowe. ParabricksOprogramowanie do genomiki, które zostało przejęte przez firmę NVIDIA w 2019 roku, jest pionierem stosu oprogramowania wykonującego różne przepływy pracy związane z analizą genomiczną za pomocą procesorów graficznych. Przetestowaliśmy Parabricksokoło dwa lata temu. Firma Dell wprowadziła wiele ulepszeń technologicznych w swoich serwerach i rozwiązaniach w zakresie pamięci masowej, a aplikacja NVIDIA Clara Parabricks wydała solidne wersje z ulepszoną akceleracją i dodaniem wywoływania wariantów. Na przykład serwer z wieloma procesorami graficznymi oparty na serwerze Dell EMC DSS 8440 z procesorami graficznymi NVIDIA® Tesla® T4 wyglądał obiecująco, jeśli chodzi o przyspieszenie dodatkowej analizy, oferując jednocześnie atrakcyjną równowagę między ceną a wydajnością. Ten blog informuje o nowej architekturze referencyjnej i wynikach testów porównawczych dla NVIDIA Clara Parabricks Analiza wtórna na serwerze DSS 8440 z wieloma procesorami graficznymi Tesla® T4 i pamięcią masową Dell Isilon F800
Architektura referencyjna
Rysunek 1 przedstawia przetestowaną architekturę referencyjną. Architektura jest modułowa i łatwa do skalowania. Seria NVIDIA Clara Parabricks Oprogramowanie używane jest za pomocą co najmniej jednego procesora graficznego, dzięki czemu skalowanie w poziomie jest maksymalnie proste. Elementy sprzętowe składają się z serwera Dell PowerEdge R640 jako węzła zarządzania, serwera DSS 8440 do obliczeń GPU oraz pamięci masowej Dell EMC Isilon F800. 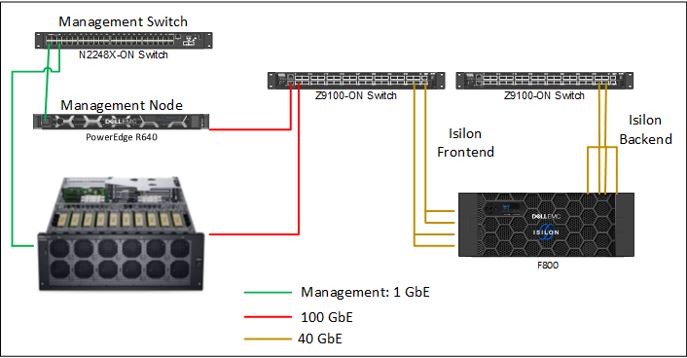
Rysunek 1. Testowana architektura referencyjna
Serwer 4U DSS 8440 z 2 gniazdami może pomieścić maksymalnie 10 wiodących w branży procesorów graficznych NVIDIA® Tesla® V100S Tensor Core, 10 procesorów GPU NVIDIA® Quadro RTX ™ lub nawet 16 procesorów graficznych NVIDIA Tesla T4, zapewniając ogromną moc obliczeniową. Szczegółową konfigurację systemu DSS 8440 przedstawiono w tabeli 1.
| Dell EMC DSS 8440 | |
|---|---|
CPU |
2x Xeon® Gold 6248R 24 cores 3.0 GHz |
RAM |
24x 64GB at 2933 MTps |
Operating System |
Red Hat Enterprise Linux Server release 7.4 (Maipo) |
BIOS System Profile |
Performance Optimized |
Logical Processor |
Disabled |
Virtualization Technology |
Disabled |
Accelerators |
16x NVIDIA® Tesla® T4 GPUs |
Parabricks |
v3.0.0.05 |
Dwa przełączniki Z9100-ON zapewniają połączenie między węzłem obliczeniowym a klastrem pamięci masowej Isilon F800. Dodatkowy przełącznik N2248X-ON służy do zarządzania.
Dane NGS
Dane do analizy porównawczej czasu wykonywania analizy wtórnej składały się z trzech zestawów danych dotyczących sekwencjonowania całego genomu ludzkiego (WGS), ERR091571


Ocena wydajności
Ulepszenia oprogramowania skracają czas pracy.
NVIDIA kontynuuje wprowadzanie ulepszeń oprogramowania do NVIDIA Clara Parabricks. Rysunek 2 przedstawia skrócenie czasu uruchomienia między dwiema wersjami Parabricks uruchamianie potoku linii zarodkowej przy użyciu środowiska testowego serwera Dell PowerEdge C4140 z 4 procesorami graficznymi V100. Przejście z wersji 2.1.0 do wersji 3.0.0 skróciło czas działania o 42%.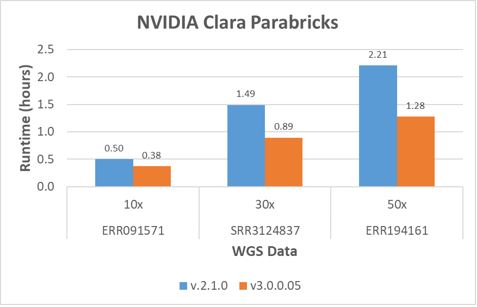
Rysunek 2: Najnowsza wersja wariantu linii zarodkowej Parabricks wywołującego środowisko uruchomieniowe potoku.
Wydajność serwera DSS 8440 z 16 procesorami T4
Czas działania NVIDIA Clara Parabricks dodatkowa analiza przy użyciu jednego procesora graficznego T4 jest o około 30% wolniejsza niż przy użyciu jednego procesora graficznego V100. Jednakże dwa (2) procesory graficzne T4 zapewniają około 10% więcej TFLOPS niż jeden (1) procesor V100 za około połowę ceny. DSS 8440 zapewnia do 16 gniazd PCIe, co otwiera możliwość zaprojektowania serwera opartego na procesorze graficznym T4, który zapewnia podobną wydajność w czasie pracy jak system C4140 z czterema procesorami graficznymi V100, ale przy niższych kosztach.
Ten Parabricks Analizę linii zarodkowej przeprowadzono przy użyciu serwera PowerEdge DSS 8440 z 16 procesorami graficznymi T4. Dla każdego przykładowego zestawu danych WGS opisanego wcześniej środowisko uruchomieniowe zostało zarejestrowane przy użyciu 1, 2, 4, 8 i 16 procesorów graficznych T4 na analizę dodatkową. Wyniki przedstawiono na rysunkach od 3 do 5. Ogólnie rzecz biorąc, czas wykonywania nie skaluje się liniowo wraz z wzrostem liczby procesorów graficznych przypadających na analizę. Wzorzec skalowania jest zbliżony do wzrostu pokrycia ilości danych na próbkę od 10x do 50x.
Chociaż nie zostało to tutaj przedstawione, wcześniejsze dochodzenie firmy Dell EMC dotyczące Parabricks Wyniki środowiska uruchomieniowego przy użyciu ośmiu lub więcej procesorów graficznych V100 na analizę nie skalowały się tak wydajnie jak w przypadku procesorów graficznych T4. Dalsze testy wykazały, że 6 procesorów graficznych T4 generowało wyniki czasu pracy prawie identyczne jak 4 procesory graficzne V100.
Rysunek 3: Porównanie wydajności z 10x WGS
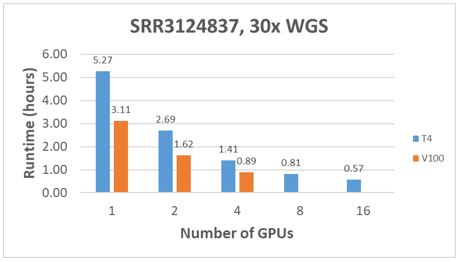
Rysunek 4: Porównanie wydajności z 30x WGS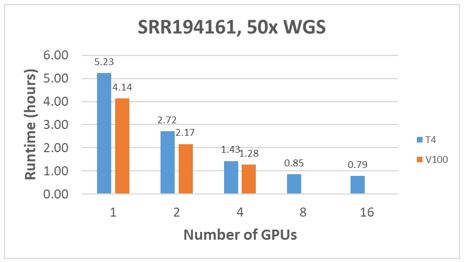
Rysunek 5: Porównanie wydajności z 50x WGS
Wnioski
DSS 8440 z szesnastoma procesorami graficznymi T4 może przetwarzać trzydzieści 50x ludzkich genomów dziennie. Podobna przepustowość codziennych analiz przy użyciu tradycyjnej architektury procesorów x86 wymaga dziesięciu węzłów obliczeniowych PowerEdge C6420. Kompletna architektura została omówiona w dokumencie "Gotowe rozwiązanie firmy Dell dla systemów HPC dla nauk przyrodniczych": Testy przepływności potoku BWA-GATK z procesorem CPU Cascade Lake i odświeżaniem Lustre ME4.
Jednak zaangażowanie wszystkich 16 procesorów graficznych T4 do przetwarzania jednej próbki przynosi niewielkie korzyści, ponieważ użycie 16 procesorów graficznych na analizę jest w najlepszym przypadku o 10% szybsze niż użycie 8 procesorów graficznych. Konstrukcja serwera DSS 8440 umożliwia równoległe wykonywanie kilku analiz wtórnych. Dzięki przydzieleniu ośmiu procesorów graficznych T4 do analizy próbki dzienna przepustowość analizy wzrasta do ~50 genomów dziennie. Użycie czterech procesorów graficznych do analizy próbki zwiększa przepustowość analizy do 70 genomów dziennie. Co ważniejsze, koszt dziennej wydajności przy użyciu procesorów graficznych T4 nie osiąga nawet połowy kosztu konstrukcji z procesorem graficznym V100.
Oprócz szybkości, dla porównywalności wyników niezbędna jest kompatybilność z innymi narzędziami analitycznymi. Polecenie Parabricks Wyniki analizy linii zarodkowej są prawie identyczne z dobrze znaną analizą haplotypu BWA-GATK z wcześniejszych testów. Chcieliśmy również porównać wyniki wywoływania wariantu Parabricks z innymi zestawami narzędzi, takimi jak samtools/mpileup. Te dwa różne narzędzia osiągają ~90% ogólnej zgodności dla zidentyfikowanych wariantów, a wariacje w wielu dobrze znanych regionach genomowych zawierających ważne geny zgadzają się ponad 99%.