Dell EMC Ready Solution for HPC Life Sciences: BWA-GATK Pipeline throughput tests with Cascade Lake CPU and Lustre ME4 Refresh
Summary: L'archiviazione come articolo si basa sulla documentazione HPC che non è più ospitata online e l'articolo non può essere modificato per soddisfare le aspettative della KB Ready Solution Dell EMC per l'HPC per le scienze della vita: BWA-GATK Pipeline throughput tests with Cascade Lake CPU and Lustre ME4 Refresh ...
Symptoms
La configurazione a 64 nodi di elaborazione delle Dell EMC Ready Solutions for HPC for Life Sciences è in grado di elaborare 194 genomi al giorno (profondità di copertura 50 volte superiore).
Panoramica
Chiamata
Lo scopo di questo blog è fornire informazioni preziose sulle prestazioni del processore Intel® Xeon® Gold 6248 per il benchmark della pipeline BWA-GATK con Dell EMC Ready Solutions for HPC Lustre Storage (aggiornamento della serie ME4) (2). La CPU Xeon® Gold 6248 è dotata di 20 core fisici o 40 core logici quando si utilizza Hyper Threading. Le configurazioni dei cluster di test sono riepilogate nella Tabella 1.
| Dell EMC PowerEdge C6420 | |
|---|---|
| CPU | 2 Xeon® Gold 6248 20 core da 2,5 GHz (Cascade Lake) |
| RAM | 12 da 16 GB a 2933 MTps |
| Sistema operativo | RHEL 7,6 |
| Interconnessione | Intel® Omni-Path |
| Profilo di sistema nel BIOS | Performance Optimized |
| Logical Processor | Disabled |
| Tecnologia di virtualizzazione | Disabled |
| BWA | 0.7.15-R1140 |
| Samtools | 1.6 |
| GATK (Italiano) | 3.6-0-g89b7209 |
I nodi di elaborazione testati sono stati collegati a Dell EMC Ready Solutions for HPC Lustre Storage tramite Intel® Omni-Path. La configurazione di riepilogo dello storage è elencata nella Tabella 2.
Tabella 2 Specifiche hardware e software della soluzione
| Dell EMC Ready Solution for Lustre Storage | |
|---|---|
| Numero di nodi | 1 Dell EMC PowerEdge R640 come Integrated Manager for Lustre (IML), 2 Dell EMC PowerEdge R740 come server di metadati (MDS), 2 Dell EMC PowerEdge R740 come server di object storage (OSS) |
| Processori | Server IML: Dual Intel Xeon Gold 5118 @ 2,3 GHz Server MDS e OSS: 2 Intel Xeon Gold 6136 a 3 GHz |
| Memoria | Server IML: 12 RDIMM DDR4 da 8 GB e 2.666 MT/s Server MDS e OSS: 24 RDIMM DDR4 da 16 GiB a 2.666 MT/s |
| Storage controller esterni |
2 HBA SAS Dell da 12 Gb/s (su ogni MDS) 4 HBA SAS Dell da 12 Gb/s (su ogni OSS) |
| Enclosure di object storage |
4 ME4084 con un totale di 336 HDD SAS NL da 8 TB a 7.200 rpm |
| Enclosure di storage dei metadati |
1 ME4024 con 24 unità SSD SAS da 960 GB. Supporta inode fino a 4,68 B |
| Controller RAID | Controller RAID SAS duplex nelle enclosure ME4084 e ME4024 |
| Sistema operativo | CentOS 7.5 x86_64 Red Hat Enterprise Linux (RHEL) 7.5 x86_64 |
| Versione del BIOS | 1.4.5 |
| Intel Omni-Path versione IFS |
10.8.0.0 |
| Versione del file system Lustre |
2.10.4 |
| Versione di IML | 4.0.7.0 |
I dati del test sono stati scelti da uno dei genomi di platino di Illumina. ERR194161 è stato elaborato con Illumina HiSeq 2000 presentato da Illumina e può essere ottenuto da EMBL-EBI. L'identificatore del DNA per questo individuo è NA12878. La descrizione dei dati dal sito web collegato mostra che questo campione ha una >profondità di copertura di 30 volte.
Valutazione delle prestazioni
Prestazioni di più nodi campione singolo
Nella Figura 1 viene riepilogato il runtime in un numero diverso di campioni e nodi di elaborazione con dati di sequenziamento dell'intero genoma (WGS) 50x. I test qui eseguiti sono progettati per dimostrare le prestazioni a livello di server, non per confronti sui singoli componenti. I data point nella Figura 1 vengono calcolati in base al numero totale di campioni, un campione per nodo di calcolo (asse X nella figura) elaborati contemporaneamente. I dettagli delle informazioni sul gasdotto BWA-GATK possono essere ottenuti dal sito web del Broad Institute (3). Il numero massimo di nodi di elaborazione utilizzati per i test è 64x C6420s. I modelli C6420s con Lustre ME4 mostrano un comportamento di dimensionamento migliore rispetto a Lustre MD3.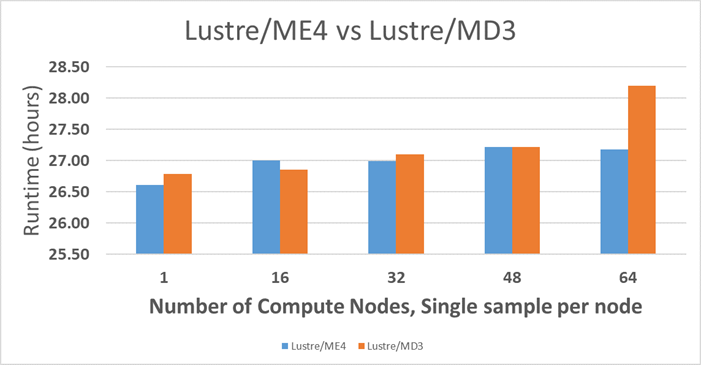
Figura 1 Confronto delle prestazioni tra Lustre MD3 e Lustre ME4
Prestazioni di più nodi campione
Un modo tipico di eseguire la pipeline NGS consiste nell'eseguire più campioni su un nodo di elaborazione e usare più nodi di calcolo per ottimizzare il throughput del processo di dati NGS. Il numero di nodi di elaborazione utilizzati per i test è 64 dei nodi di elaborazione C6420 e il numero di campioni per nodo è di cinque campioni. Fino a 320 campioni vengono elaborati contemporaneamente per stimare il numero massimo di genomi al giorno senza errori di processo.Come illustrato nella Figura 2, un singolo nodo di elaborazione C6420 può elaborare 3,24 dei 50 genomi umani interi al giorno quando vengono processati contemporaneamente 5 campioni. Per ogni campione, vengono allocati 7 core e 30 GB di memoria.
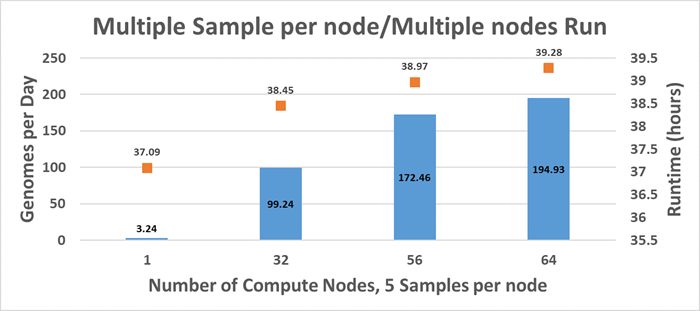
Figura 2 Test di throughput con un massimo di 64 modelli C6420 e Lustre ME4
320 genomi umani completi su 50 possono essere elaborati con 64 nodi di elaborazione C6420 in 40 ore. In altre parole, le prestazioni della configurazione di test si riepilogano in 194 genomi al giorno per l'intero genoma umano con una profondità di copertura di 50 volte.
Conclusione
Poiché la dimensione dei dati di WGS è in costante crescita. L'attuale dimensione media di WGS è 50x. Si tratta di un valore 5 volte superiore a quello di un tipico WGS di 4 anni fa, quando abbiamo iniziato a confrontare la pipeline BWA-GATK. L'aumento dei dati non grava sulla capacità lato storage poiché la maggior parte delle applicazioni nella pipeline è limitata anche dalla velocità di clock della CPU. Di conseguenza, con l'aumento delle dimensioni dei dati, la pipeline viene eseguita più a lungo anziché generare più scritture.Tuttavia, durante il processo viene generato un numero maggiore di file temporanei a causa della maggiore quantità di dati da parallelizzare e questo numero maggiore di file temporanei aperti contemporaneamente esaurisce il limite di file aperti in un sistema operativo Linux. Una delle applicazioni non viene completata in modo invisibile all'utente raggiungendo il limite del numero di file aperti. Una soluzione semplice consiste nell'aumentare il limite a >150K.
Tuttavia, la Ready Solution con Lustre ME4 come spazio scratch ha una capacità di throughput migliore rispetto alla versione precedente. Ora, 64 nodi Ready Solution segna 194 genomi al giorno di potenza di elaborazione per un livello di lavoro 50 volte superiore.
Risorse
1. Un'indagine sugli strumenti per l'analisi delle varianti dei dati di sequenziamento del genoma di nuova generazione. Pabinger S, Dander A, Fischer M, Snajder R, Sperk M, Efremova M, Krabichler B, Speicher MR, Zschocke J, Trajanoski Z. 2, s.l. : Brief Bioinform, marzo 2014, vol. 15 (2). 10.1093/bib/bbs086.2. Dell EMC Ready Solution for HPC Lustre Storage. (Articolo non più disponibile per riferimento, estratto dal team HPC)
3. Toolkit per l'analisi del genoma. https://software.broadinstitute.org/gatk/

Cause
L'archiviazione come articolo si basa sulla documentazione HPC che non è più ospitata online e l'articolo non può essere modificato per soddisfare le aspettative della KB
Resolution
L'archiviazione come articolo si basa sulla documentazione HPC che non è più ospitata online e l'articolo non può essere modificato per soddisfare le aspettative della KB