PowerEdge: Podpora genomického sekvencování pomocí kanálu FAGP (Falcon Accelerated Genomics Pipeline) na kartě Intel IFPGA PAC
Summary: Systém Falcon Accelerated Genomics Pipeline s jedinou programovatelnou akcelerační kartou Intel FPGA dokáže zpracovat 50násobek celých lidských genomů za méně než 3 hodiny prostřednictvím kanálu Alternative Variant Calling Pipeline. ...
Symptoms
Systém Falcon Accelerated Genomics Pipeline s jedinou programovatelnou akcelerační kartou Intel FPGA dokáže zpracovat 50násobek celých lidských genomů za méně než 3 hodiny prostřednictvím kanálu Alternative Variant Calling Pipeline.
Přehled, výzva trhu (potřeba), řešení Falcon odpovídá na potřebu:
Přesná medicína, genomika a epigenetika využívají sekvenování genomu k provádění výzkumu, zlepšování diagnostiky, vývoji léčiv, zvyšování kvality péče pro poskytovatele zdravotní péče a optimalizaci rostlinné výroby. Pro vědy o živé přírodě je nyní analýza genomu klíčovou aplikací, částečně kvůli velkému snížení nákladů na sběr dat díky pokroku v sekvenování nové generace (NGS). Kromě zvýšeného sběru dat došlo také k významnému nárůstu rozsahu genomických aplikací používaných napříč univerzitami, centry genomického výzkumu, farmaceutickými společnostmi a zdravotnickými organizacemi.
Každých sedm měsíců se množství genomových dat zdvojnásobuje (1). Efektivní a nákladově efektivní zpracování dat se stalo kritickým. Výpočetní výkon řešení pouze s procesory se neškáluje dostatečně rychle, aby udržel krok s růstem genomických dat. To vedlo k potřebě hardwarové akcelerace. Akcelerátory, jako jsou FPGA, se stávají klíčovými při plnění výpočetních požadavků této exploze genomických dat. Ve srovnání s jinými hardwarově akcelerovanými řešeními nabízí Falcon Accelerated Genomics Pipeline (FAGP) flexibilitu, vysokou propustnost a nižší náklady na vzorek.
Co je FPGA, nabídka Intel PAC a výhoda:
FPGA jsou křemíková zařízení, která lze dynamicky přeprogramovat pomocí datové cesty, která přesně odpovídá vašim úlohám, jako je sekvenování genomu, analýza dat nebo komprese, jak je znázorněno na obrázku 1. Tato všestrannost umožňuje poskytovat rychlejší výpočetní výkon, energeticky úspornější výpočty a služby s nižší latencí, což snižuje celkové náklady na vlastnictví a maximalizuje výpočetní kapacitu v rámci výkonových, prostorových a chladicích omezení datových center.
FPGA tradičně vyžadují k programování hluboké odborné znalosti domény. Kvůli zjednodušení vývoje a umožnění rychlého nasazení v datovém centru nabízí společnost Intel akcelerační platformu, která obsahuje programovatelné akcelerační karty Intel FPGA (Intel FPGA PAC) založené na PCI Express* (PCIe*) a akcelerační sadu Intel® pro procesory Intel Xeon® s FPGA. Tyto platformy Intel jsou kvalifikované, ověřené a nasazené prostřednictvím společnosti Dell EMC. Společně s partnery ekosystému, jako je Falcon Computing, nabízí akcelerační platforma Intel spolehlivé a připravené řešení s transparentním hardwarem pod kapotou.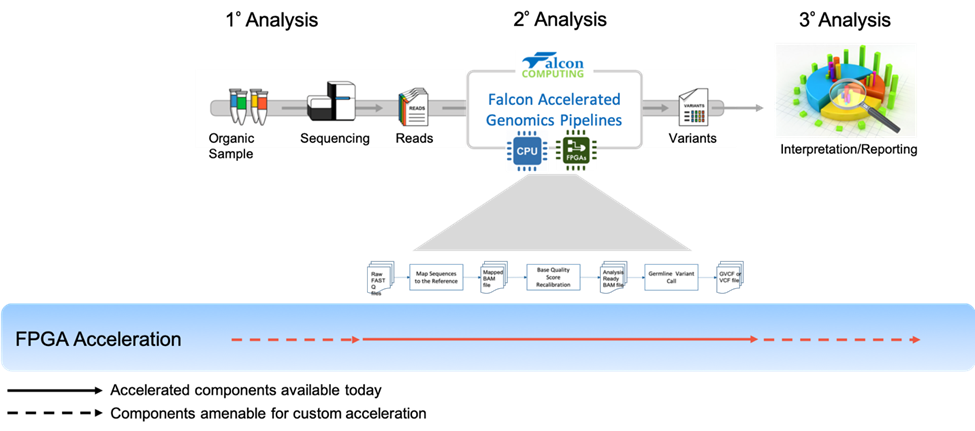
Obrázek 1: Vylepšená přesnost a rychlost standardního kanálu GATK
Podrobnosti o řešení Falcon:
Sada nástrojů pro analýzu genomu (GATK) je zlatým standardem pro zpracování genomických dat přijímaným komunitou genomiky (2). Jeho Best Practice Workflow (BPW) je známý svou pomalostí při výpočtech při generování výsledků pro velké vzorky, jako je Whole-Genome (WGS). K vyřešení tohoto problému vyvinula společnost Falcon Computing Solutions flexibilní softwarový balíček nástrojů, který se řídí BPW a lze jej implementovat na více platformách a architekturách. Ve srovnání s kanály GATK založenými na procesoru je rychlý o několik řádů.
FAGP poskytuje komplexní řešení pro nákladově efektivní analýzu genomických dat pomocí kanálu GATK s vysokým výkonem, přesností a reprodukovatelností. Řešení poskytuje až 15násobné zrychlení se stejnou přesností jako GATK (3). To znamená, že analýza, která obvykle trvá 50 až 60 hodin, může být provedena za méně než 4 hodiny (3). FAGP poskytuje výjimečnou úroveň akcelerace a přesnosti díky výkonným, spolehlivým FPGA Intel Arria 10 a procesorům Intel® Xeon®.
FAGP následuje GATK BPW. Implementuje akceleraci v mnoha komponentách kanálů od zarovnání (BWA) po volání variant (HaplotypeCaller) (4). Kromě akcelerovaného BWA obsahuje také akcelerovanou verzi aligneru Minimap2, který je součástí Alternate Genomic Pipeline od společnosti Falcon (5). Alternativní kanál poskytuje ještě rychlejší řešení. Dokáže dokončit 50x sekvenování celého genomu během 3 hodin. Obě rovnátka mají funkci generování označených duplikátů a seřazených čtení bez nutnosti použití dalších nástrojů.
FAGP dosahuje vysokého výkonu/propustnosti zrychlením intenzivních výpočtů v kanálu GATK pomocí platforem Intel FPGA PAC. To se liší od řešení se škálováním na více systémů, která dosahují vysoké propustnosti přidáním dalších prostředků procesoru. Taková škálovaná řešení mají omezenou schopnost snížit náklady nebo latenci na vzorek.
Další výhodou řešení Falcon je, že se jedná o otevřený plynovod jako GATK. Uživatelé mohou ovládat jednotlivé kroky v potrubí. Mezilehlá data jsou uložena a lze k nim přistupovat.
Tabulka 1: Výhody Falcon Accelerated Genomics Pipeline
| Výhody FAGP (Falcon Accelerated Genomics Pipeline) | |
|---|---|
| Skutečný GATK | Podpora více verzí GATK, včetně 4.0 |
| Průmyslové měřítko | Spusťte pět celých genomů nebo 24 celých exomů za jeden den. |
| Alternativní varianta | < 3hodinová doba vyřízení na pracovišti pro WGS (50X) |
| Rychlost | Provádějte kanály s osvědčenými postupy GATK až >15krát rychleji. |
| Využití stávajících | Není třeba přepisovat pracovní algoritmy. |
Konfigurace hardwaru Dell
Tabulka 2: Dell EMC PowerEdge R740xd jako testovací zařízení
| Dell EMC PowerEdge R740xd | |
|---|---|
| Procesor | 2x Intel(R) Xeon(R) Gold 6148 CPU @ 2.40GHz |
| Paměť | 384GB @ 32x 16GB RDIMM, 2666MT/s, Dual Rank |
| Úložiště | 4x 1.2TB 10K RPM SAS 12Gbps 512n 2.5in Hot-plug Hard Drive in RAID 0 2x INTEL SSDPEDMD020T4 DC P3700 1.8T in software RAID 0 |
| FPGA | Intel Programmable Acceleration Card with Intel Arria® 10 GX FPGA (Intel Acceleration Stack 1.1) |
| Profil systému | Performance |
| Verze systému BIOS | 2.1.3 |
| Hyperthreading | Enabled |
| Operační systém | Red Hat Enterprise Linux Server release 7.4 (Maipo) (3.10.0-693.el7.x86_64) |
Vyhodnocení výkonu
V našem srovnávacím testování jsme použili data sekvenování celého lidského genomu s 10x, 30x a 50x hloubkou pokrytí.
Tabulka 3: Testovaná celogenomová sekvenační data
| Spustit přistoupení | Hloubka pokrytí | Datový spoj |
|---|---|---|
| ERR091571 | 10ks | https://www.ebi.ac.uk/ena/data/view/ERR091571 |
| SRR3124837 | 30ks | https://www.ebi.ac.uk/ena/data/view/SRR3124837 |
| ERR194161 | 50x | https://www.ebi.ac.uk/ena/data/view/ERR194161 |
Výsledky:
Tabulka 4 shrnuje čas potřebný k dokončení procesu vzorových postupů GATK 4.0 během tří testovacích cyklů pomocí FAGP a Intel FPGA PAC v serveru DELL EMC PowerEdge R740xd.
Tabulka 4: Celkový počet modulů runtime z kanálu osvědčených postupů verze 2.1.1
| Ukázka | Hloubka pokrytí | Test 1 | Doba trvání (minuty) Test 2 |
Test 3 |
|---|---|---|---|---|
ERR091571 |
10x |
75.63 |
76.67 |
76.38 |
SRR3124837 |
30x |
160.00 |
162.77 |
161.38 |
ERR194161 |
50x |
242.97 |
250.65 |
247.18 |
Tabulka 5 shrnuje čas (v minutách) potřebný k dokončení alternativního kanálu: Zárodečná linie Falcon během tří testovacích cyklů pomocí nástroje FAGP a Intel FPGA PAC v serveru DELL EMC PowerEdge R740xd.
Tabulka 5: Celkový počet modulů runtime z kanálu volání alternativních variant
| Ukázka | Hloubka pokrytí | Test 1 | Doba trvání (minuty) Test 2 |
Test 3 |
|---|---|---|---|---|
ERR091571 |
10x |
62.70 |
58.21 |
59.80 |
SRR3124837 |
30x |
130.38 |
129.90 |
129.95 |
ERR194161 |
50x |
171.52 |
171.87 |
171.37 |
Shrnutí řešení Falcon Genomic Solution
Falcon Accelerated Genomics Pipeline nabízí vysokou propustnost a nízké náklady/vzorek/den. Společně s programovatelnou akcelerační kartou Intel FPGA a certifikovaným serverem DELL poskytuje FAGP kompletní řešení, které lze použít pro aplikace sekvenování genomu."
V TCGB poskytujeme služby sekvenování genomu našim klientům po celé zemi. Falcon Accelerated Genomics Pipeline* nám umožnil zkrátit obrat ze dnů na několik hodin při zachování přesnosti průmyslových standardů GATK pipelines."
— Dr. Xinmin Li, ředitel Technologického centra pro genomiku a bioinformatiku (TCGB) UCLA
Zdroje
1. Sekvenováním genomu vzniká tolik dat, že nevíme, co s nimi. [Online] https://www.washingtonpost.com/news/speaking-of-science/wp/2015/07/07/sequencing-the-genome-creates-so-much-data-we-don't-know-what-to-do-with-it.
2. GATK [Online] https://software.broadinstitute.org/gatk/
3. Akcelerovaná genomika [online] http://www.falconcomputing.com/falcon-accelerated-genomics-pipeline
4. BWA. [Online]
http://bio-bwa.sourceforge.net/bwa.shtml5. Minimapa2. [Online] https://github.com/lh3/minimap2