PowerEdge: Øk genomikksekvensialisering med Falcon Accelerated Genomics Pipeline (FAGP) på Intel FPGA PAC
Summary: Falcon Accelerated Genomics Pipeline med et enkelt Intel FPGA Programmable Acceleration Card kan behandle 50x hele menneskelige genomer på mindre enn 3 timer gjennom Alternative Variant Calling Pipeline. ...
Symptoms
Falcon Accelerated Genomics Pipeline med et enkelt Intel FPGA Programmable Acceleration Card kan behandle 50x hele menneskelige genomer på mindre enn 3 timer gjennom Alternative Variant Calling Pipeline.
Oversikt, markedsutfordring (behov), Falcon løsning svarer på behovet:
Presisjonsmedisin, genomikk og epigenetikk bruker genomisk sekvensering for å utføre forskning, forbedre diagnose, utvikle legemidler, øke kvaliteten på omsorg for helsepersonell og optimalisere avlinger. For biovitenskap er genomanalyse nå en nøkkelapplikasjon, delvis på grunn av den store kostnadsreduksjonen av datainnsamling fra fremskritt innen neste generasjons sekvensering (NGS). I tillegg til økt datainnsamling har det også vært betydelig vekst i utvalget av genomiske applikasjoner som brukes på tvers av universiteter, genomiske forskningssentre, farmasøytiske selskaper og helseorganisasjoner.
Hver syvende måned dobles mengden genomdata (1). Databehandling på en effektiv og kostnadseffektiv måte har blitt kritisk. Beregningskraften til rene prosessorløsninger skaleres ikke raskt nok til å holde tritt med veksten i genomiske data. Dette har ført til behovet for maskinvareakselerasjon. Akseleratorer som FPGA-er blir avgjørende for å matche beregningskravene til denne genomiske dataeksplosjonen. Sammenlignet med andre maskinvareakselererte løsninger, tilbyr Falcon Accelerated Genomics Pipeline (FAGP) fleksibilitet, høy gjennomstrømning og lavere kostnad per prøve.
Hva er FPGA, Intel PAC-tilbud og fordel:
FPGA-er er silisiumenheter som kan omprogrammeres dynamisk med en databane som samsvarer nøyaktig med arbeidsbelastningene dine, for eksempel genomsekvensering, dataanalyse eller komprimering som illustrert i figur 1. Denne allsidigheten muliggjør raskere behandling, mer strømeffektiv beregning og service med lavere ventetid – noe som reduserer de totale eierkostnadene og maksimerer databehandlingskapasiteten innenfor begrensningene for strøm, plass og kjøling for datasentrene dine.
Tradisjonelt krever FPGA-er dyp domeneekspertise for å programmere. For å forenkle utviklingsflyten og muliggjøre rask distribusjon på tvers av datasenteret, tilbyr Intel en akselerasjonsplattform som inkluderer PCI Express* (PCIe*)-baserte Intel FPGA programmerbare akselerasjonskort (Intel FPGA PAC) og Intel® Acceleration Stack for Intel Xeon® CPU med FPGA-er. Disse Intel-plattformene er kvalifiserte, validerte og distribuert gjennom Dell EMC. Sammen med økosystempartnere som Falcon Computing tilbyr Intel Acceleration Platform en pålitelig og bruksklar løsning med gjennomsiktig maskinvare under panseret.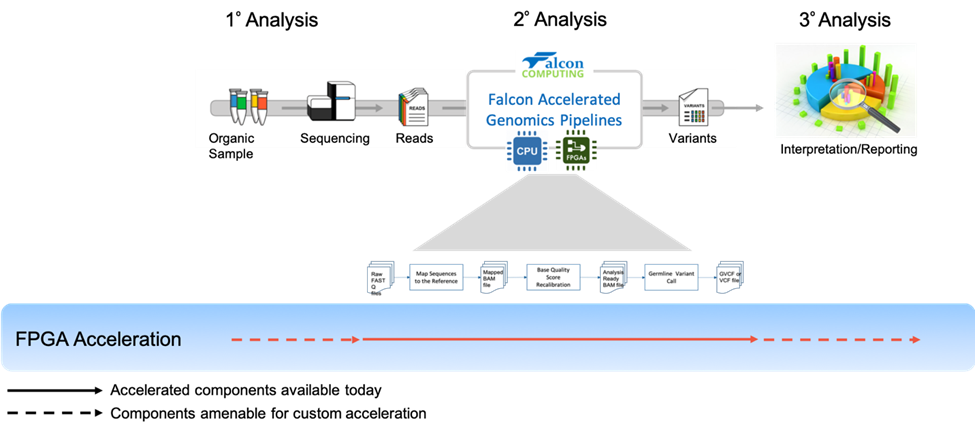
Figur 1: Forbedret nøyaktighet og hastighet på standard GATK-datasamlebånd
Detaljer om Falcon-løsningen:
Genome Analysis Toolkit (GATK) er gullstandarden for genomisk databehandling akseptert av genomikkmiljøet (2). Best Practice Workflow (BPW) er kjent for sin treghet i beregning for å generere resultater for store prøver som Whole-Genome (WGS). For å løse dette problemet har Falcon Computing Solutions utviklet en fleksibel programvarepakke med verktøy som følger BPW og kan implementeres i flere plattformer og arkitekturer. Det er raskt med flere størrelsesordener sammenlignet med CPU-baserte GATK-rørledninger.
FAGP tilbyr en ende-til-ende-løsning for kostnadseffektiv analyse av genomiske data ved hjelp av GATK-pipelinen med høy ytelse, nøyaktighet og reproduserbarhet. Løsningen leverer opptil 15x speedup med samme nøyaktighet som GATK (3). Det betyr at en analyse som typisk tar 50 til 60 timer, kan gjennomføres på under 4 timer (3). FAGP gir eksepsjonelle nivåer av akselerasjon og nøyaktighet med høy ytelse, pålitelige Intel Arria 10 FPGA-er og Intel® Xeon-prosessorer®.
FAGP følger GATK BPW. Den implementerer akselerasjon i mange komponenter i rørledningene fra justering (BWA) til variantkall (HaplotypeCaller) (4). I tillegg til den akselererte BWA-en inkluderer den også en akselerert versjon av Minimap2 som er en del av Alternate Genomic Pipeline fra Falcon (5). Den alternative pipelinen gir en enda raskere løsning. Den kan fullføre 50x helgenomsekvensering innen 3 timer. Begge justeringene har funksjonen for å generere merkede duplikater og sorterte lesninger uten å måtte bruke tilleggsverktøy.
FAGP oppnår høy ytelse/gjennomstrømning ved å akselerere intensiv databehandling i GATK-pipelinen ved hjelp av Intel FPGA PAC-plattformer. Dette er forskjellig fra utskalerte løsninger som oppnår høy gjennomstrømning ved å legge til flere CPU-ressurser. Slike utskalerte løsninger har begrenset evne til å redusere kostnader eller ventetid per prøve.
En annen fordel med Falcon-løsningen er at det er en åpen rørledning som GATK. Brukere kan kontrollere enkelttrinn i pipelinene. Mellomliggende data lagres og kan nås.
Tabell 1 Fordeler med Falcon Accelerated Genomics Pipeline
| Fordeler med Falcon Accelerated Genomics Pipeline (FAGP) | |
|---|---|
| Ekte GATK | Støtte for flere GATK-versjoner, inkludert 4.0 |
| Bransjeskala | Kjør fem hele genomer eller 24 hele eksomer på en dag. |
| Alternativ variant | < 3-timers behandlingstid på stedet for WGS (50X) |
| Speed (Hastighet) | Utfør planlegging for >anbefalte fremgangsmåter for GATK opptil 15 ganger raskere. |
| Dra nytte av eksisterende | Du trenger ikke å omskrive arbeidsalgoritmer. |
Dell-maskinvarekonfigurasjon
Tabell 2: Dell EMC PowerEdge R740xd som testmiljø
| Dell EMC PowerEdge R740xd | |
|---|---|
| Prosessor | 2x Intel(R) Xeon(R) Gold 6148 CPU @ 2.40GHz |
| Minne | 384GB @ 32x 16GB RDIMM, 2666MT/s, Dual Rank |
| Lagring | 4x 1.2TB 10K RPM SAS 12Gbps 512n 2.5in Hot-plug Hard Drive in RAID 0 2x INTEL SSDPEDMD020T4 DC P3700 1.8T in software RAID 0 |
| FPGA | Intel Programmable Acceleration Card with Intel Arria® 10 GX FPGA (Intel Acceleration Stack 1.1) |
| Systemprofil | Performance |
| BIOS-versjon | 2.1.3 |
| Hyperthreading | Enabled |
| OPERATIVSYSTEM | Red Hat Enterprise Linux Server release 7.4 (Maipo) (3.10.0-693.el7.x86_64) |
Ytelsesevaluering
I vår benchmark-testing brukte vi hele menneskelige genomsekvenseringsdata med 10x, 30x og 50x dybde av dekning.
Tabell 3: Testet helgenomsekvenseringsdata
| Kjør tiltredelse | Dekningsdybde | Datakobling |
|---|---|---|
| ERR091571 | 10x | https://www.ebi.ac.uk/ena/data/view/ERR091571 |
| SRR3124837 | 30x | https://www.ebi.ac.uk/ena/data/view/SRR3124837 |
| ERR194161 | 50x | https://www.ebi.ac.uk/ena/data/view/ERR194161 |
Resultater:
Tabell 4 oppsummerer tiden det tok å fullføre planleggingen av beste praksis for GATK 4.0 over tre testsykluser med FAGP og Intel FPGA PAC som ligger i DELL EMC PowerEdge R740xd-serveren.
Tabell 4: Total kjøretid fra Best Practice Pipeline versjon 2.1.1
| Eksempel | Dekningsdybde | Test 1 | Kjøretid (minutter) Test 2 |
Test 3 |
|---|---|---|---|---|
ERR091571 |
10x |
75.63 |
76.67 |
76.38 |
SRR3124837 |
30x |
160.00 |
162.77 |
161.38 |
ERR194161 |
50x |
242.97 |
250.65 |
247.18 |
Tabell 5 oppsummerer tiden (i minutter) det tar å fullføre den alternative pipelinen: Falcon Germline over tre testsykluser ved hjelp av FAGP og Intel FPGA PAC som er plassert i DELL EMC PowerEdge R740xd-serveren.
Tabell 5: Total kjøretid fra alternativ variantkallingspipeline
| Eksempel | Dekningsdybde | Test 1 | Kjøretid (minutter) Test 2 |
Test 3 |
|---|---|---|---|---|
ERR091571 |
10x |
62.70 |
58.21 |
59.80 |
SRR3124837 |
30x |
130.38 |
129.90 |
129.95 |
ERR194161 |
50x |
171.52 |
171.87 |
171.37 |
Sammendrag av Falcon Genomic Solution
Falcon Accelerated Genomics Pipeline tilbyr høy gjennomstrømning, lav kostnad / prøve / dag fordel. Sammen med Intel FPGA Programmable Acceleration Card og den sertifiserte DELL-serveren tilbyr FAGP en komplett løsning som kan tas i bruk for dine genomsekvenseringsapplikasjoner."
Hos TCGB leverer vi genomsekvenseringstjenester til våre landsdekkende kunder. Falcon Accelerated Genomics Pipeline* har gjort det mulig for oss å kutte behandlingstiden fra dager til noen få timer, samtidig som vi opprettholder nøyaktigheten til industristandardiserte GATK-rørledninger."
- Dr. Xinmin Li, direktør for teknologisenter for genomikk og bioinformatikk (TCGB) UCLA
Ressurser
1. Sekvensering av genomet skaper så mye data at vi ikke vet hva vi skal gjøre med det. [På nett] https://www.washingtonpost.com/news/speaking-of-science/wp/2015/07/07/sequencing-the-genome-creates-so-much-data-we-don't-know-what-to-do-with-it.
2. GATK [På nett] https://software.broadinstitute.org/gatk/
3. Akselerert genomikk [Online] http://www.falconcomputing.com/falcon-accelerated-genomics-pipeline
4. BWA. [På nett]
http://bio-bwa.sourceforge.net/bwa.shtml5. Minikart2. [På nett] https://github.com/lh3/minimap2