Dell EMC Ready Solution для НРС в области медико-биологических наук. Тесты пропускной способности методики BWA–GATK с ЦП Cascade Lake и обновлением Lustre ME4
Summary: Архивирование в виде статьи основано на документации по HPC, которая больше не размещена в Интернете, и статью нельзя отредактировать, чтобы она соответствовала ожиданиям базы знаний Готовое решение Dell EMC для HPC медико-биологических наук: Тесты пропускной способности методики BWA–GATK с ЦП Cascade Lake и обновлением Lustre ME4 ...
Symptoms
Конфигурация с 64 вычислительными узлами решений Dell EMC Ready Solutions для HPC медико-биологических наук может обрабатывать 194 генома в день (в 50 раз больше глубины покрытия).
Обзор
Вызов
Цель этого блога — предоставить ценную информацию о производительности процессора Intel® Xeon® Gold 6248 для эталонного тестирования конвейера BWA-GATK с помощью решений Dell EMC Ready Solutions для хранилища данных HPC Lustre (обновление серии ME4) (2). Процессор Xeon® Gold 6248 имеет 20 физических ядер или 40 логических ядер при использовании технологии Hyper-Threading. Конфигурации тестовых кластеров приведены в Таблице 1.
| Dell EMC PowerEdge C6420 | |
|---|---|
| ЦП | 2 процессора Xeon® Gold 6248, 20 ядер, 2,5 ГГц (Cascade Lake) |
| RAM | 12 модулей памяти емкостью 16 Гбайт при 2933 млн транзакций в секунду |
| ОС | RHEL 7.6 |
| Соединение | Intel® Omni-Path |
| Системный профиль BIOS | Оптимизация производительности |
| Логический процессор | Disabled |
| Технология виртуализации | Disabled |
| БДЖ | 0.7.15 — Р1140 |
| Самтулс | 1.6 |
| ГАТК | 3.6-0-Г89Б7209 |
Протестированные вычислительные узлы были подключены к готовым решениям Dell EMC для хранилища данных HPC Lustre с помощью технологии Intel® Omni-Path. Сводная конфигурация хранилища приведена в Таблице 2.
Таблица 2. Технические характеристики аппаратного и программного обеспечения решения
| Готовое решение Dell EMC для хранилища Lustre | |
|---|---|
| Количество узлов | 1 сервер Dell EMC PowerEdge R640 в качестве интегрированного диспетчера для Lustre (IML) 2 сервера Dell EMC PowerEdge R740 в качестве сервера метаданных (MDS) 2 сервера Dell EMC PowerEdge R740 в качестве сервера объектного хранилища (OSS) |
| Процессоры | Сервер IML. Два Intel Xeon Gold 5118 @ 2.3 ГГц Серверы MDS и OSS: Два процессора Intel Xeon Gold 6136 на 3,00 ГГц |
| Память | Сервер IML. 12 модулей DDR4 RDIMM емкостью 8 Гбайт, 2666 млн транзакций в секунду Серверы MDS и OSS: 24 модуля DDR4 RDIMM 16 Гбайт, 2 666 млн транзакций в секунду |
| Внешние контроллеры системы хранения данных |
2 адаптера главной шины Dell SAS 12 Гбит/с (в каждом MDS) 4 адаптера главной шины Dell SAS 12 Гбит/с (в каждом OSS) |
| Шасси объектных систем хранения |
4 накопителя ME4084 с 336 жесткими дисками SAS NL емкостью 8 Тбайт, 7200 об/мин |
| Шасси хранилища метаданных |
1 накопитель ME4024 с 24 твердотельными накопителями SAS емкостью 960 Гбайт. Поддержка индексных дескрипторов до 4,68 млрд |
| RAID-контроллеры | Дуплексные RAID-контроллеры SAS в полках ME4084 и ME4024 |
| Операционная система | CentOS 7.5 x86_64 Red Hat Enterprise Linux (RHEL) 7.5 x86_64 |
| Версия BIOS | 1.4.5 |
| Версия Intel Omni-Path IFS |
10.8.0.0 |
| Версия файловой системы Lustre |
2.10.4 |
| Версия IML | 4.0.7.0 |
Тестовые данные были выбраны из одного из платиновых геномов Illumina. ERR194161 был обработан с помощью Illumina HiSeq 2000, представленного компанией Illumina, и может быть получен от EMBL-EBI. Идентификатором ДНК этого человека является NA12878. Описание данных с веб-сайта, на который дается ссылка, показывает, что этот образец имеет >30-кратную глубину покрытия.
Оценка производительности
Производительность одного образца нескольких узлов
На рисунке 1 показана сумма времени выполнения в различных образцах и вычислительных узлах с 50-кратным полногеномным секвенированием (WGS). Проводимые здесь тесты предназначены для демонстрации производительности на уровне сервера, а не для сравнения отдельных компонентов. Точки данных на рисунке 1 вычисляются на основе общего количества выборок, по одной выборке на вычислительный узел (ось X на рисунке), которые обрабатываются одновременно. Подробную информацию о трубопроводе BWA-GATK можно получить на веб-сайте Института Броуда (3). Максимальное количество вычислительных узлов, используемых для тестирования, составляет 64 x C6420. C6420 с Lustre ME4 демонстрируют лучшее поведение при масштабировании, чем Lustre MD3.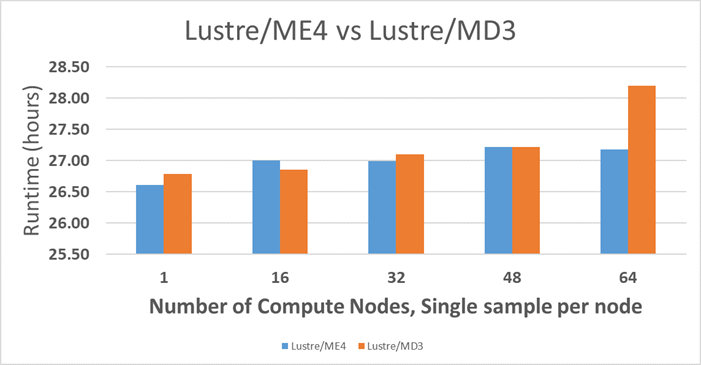
Рис. 1. Сравнение производительности Lustre MD3 и Lustre ME4
Производительность нескольких образцов нескольких узлов
Типичный способ запуска конвейера NGS заключается в выполнении нескольких выборок на вычислительном узле и использовании нескольких вычислительных узлов для максимального увеличения пропускной способности обработки данных NGS. Для тестов используется 64 вычислительных узла C6420, а количество выборок на узел — 5. До 320 образцов обрабатываются одновременно, чтобы оценить максимальное количество геномов в день без сбоев в работе.Как показано на рис. 2, один вычислительный узел C6420 может обрабатывать 3,24 из 50 целых геномов человека в день при одновременной обработке 5 образцов. Для каждого образца выделяется 7 ядер и 30 ГБ памяти.
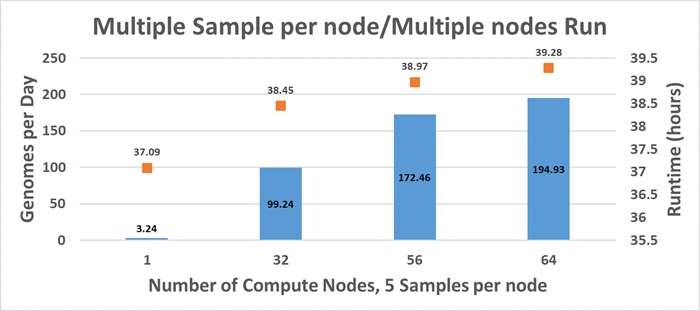
Рис. 2 Тесты пропускной способности с использованием до 64 C6420 и Lustre ME4
320 из 50x целых геномов человека могут быть обработаны с помощью 64 вычислительных узлов C6420 за 40 часов. Другими словами, производительность тестовой конфигурации составляет 194 генома в день для всего генома человека с 50-кратной глубиной покрытия.
Заключение
Поскольку объем данных WGS постоянно растет. Текущий средний размер WGS составляет 50x. Это в 5 раз больше, чем типичный WGS 4 года назад, когда мы начинали тестировать трубопровод BWA-GATK. Увеличение объема данных не приводит к увеличению емкости на стороне хранилища, поскольку большинство приложений в конвейере также ограничены тактовой частотой ЦП. Таким образом, с увеличением объема данных конвейер выполняется дольше, а не генерирует больше операций записи.Однако в ходе процесса создается большее количество временных файлов из-за того, что больше данных нужно распараллелить, и это увеличение количества временных файлов, открываемых одновременно, исчерпывает лимит открытых файлов в операционной системе Linux. Одно из приложений не завершается без уведомления о сбое из-за достижения предельного количества открытых файлов. Простое решение — увеличить лимит до >150К.
Тем не менее, Ready Solution с Lustre ME4 в качестве вспомогательного пространства имеет более высокую пропускную способность, чем предыдущая версия. Теперь 64 узла Ready Solution отмечают вычислительную мощность 194 генома в день для 50x WGS.
Ресурсы
1. Обзор инструментов для вариантного анализа данных секвенирования генома нового поколения. Пабингер С., Дандер А., Фишер М., Снайдер Р., Шперк М., Ефремова М., Крабихлер Б., Шпайхер М.Р., Цшоке Й., Траяноски З. 2, с.л. . Бриф Биоинформ, март 2014, том 15 (2). 10.1093/нагрудник/bbs086.2. Готовое решение Dell EMC для хранилищ НРС Lustre. (Статья больше недоступна для справки, ее получила команда HPC)
3. Набор инструментов для анализа генома. https://software.broadinstitute.org/gatk/

Cause
Архивирование в виде статьи основано на документации по HPC, которая больше не размещена в Интернете, и статью нельзя отредактировать, чтобы она соответствовала ожиданиям базы знаний
Resolution
Архивирование в виде статьи основано на документации по HPC, которая больше не размещена в Интернете, и статью нельзя отредактировать, чтобы она соответствовала ожиданиям базы знаний