PowerEdge: Incrementare il sequenziamento genomico con Falcon Accelerated Genomics Pipeline (FAGP) in Intel FPGA PAC (in inglese)
Summary: Falcon Accelerated Genomics Pipeline con una singola scheda di accelerazione programmabile Intel FPGA è in grado di elaborare genomi umani interi 50 volte superiori in meno di 3 ore tramite Alternative Variant Calling Pipeline. ...
Symptoms
Falcon Accelerated Genomics Pipeline con una singola scheda di accelerazione programmabile Intel FPGA è in grado di elaborare genomi umani interi 50 volte superiori in meno di 3 ore tramite Alternative Variant Calling Pipeline.
Panoramica, Sfida di mercato (necessità), La soluzione Falcon risponde all'esigenza:
La medicina di precisione, la genomica e l'epigenetica utilizzano il sequenziamento genomico per condurre ricerche, migliorare la diagnosi, sviluppare farmaci, aumentare la qualità delle cure per gli operatori sanitari e ottimizzare la produzione agricola. Per le scienze della vita, l'analisi del genoma è ora un'applicazione chiave, in parte grazie alla notevole riduzione dei costi di raccolta dei dati grazie ai progressi nel sequenziamento di nuova generazione (NGS). Oltre all'aumento della raccolta di dati, si è registrata anche una crescita significativa della gamma di applicazioni genomiche utilizzate in università, centri di ricerca genomica, aziende farmaceutiche e organizzazioni sanitarie.
Ogni sette mesi la quantità di dati sul genoma raddoppia (1). L'elaborazione dei dati in modo efficiente ed economico è diventata fondamentale. La potenza di elaborazione delle soluzioni basate solo su processore non sta crescendo abbastanza velocemente da stare al passo con la crescita dei dati genomici. Ciò ha generato la necessità di accelerare l'hardware. Gli acceleratori come gli FPGA stanno diventando fondamentali per soddisfare le esigenze computazionali di questa esplosione dei dati genomici. Rispetto ad altre soluzioni accelerate dall'hardware, la Falcon Accelerated Genomics Pipeline (FAGP) offre flessibilità, produttività elevata e un costo inferiore per campione.
Che cos'è FPGA, l'offerta e il vantaggio di Intel PAC:
Gli FPGA sono dispositivi al silicio che possono essere riprogrammati dinamicamente con un percorso dati che corrisponde esattamente ai carichi di lavoro, come il sequenziamento genomico, l'analisi dei dati o la compressione, come illustrato nella Figura 1. Questa versatilità consente il provisioning di un elaborazione più rapida, un calcolo più efficiente dal punto di vista energetico e un servizio a bassa latenza, riducendo i costi complessivi di gestione e ottimizzando la capacità di elaborazione entro i limiti di alimentazione, spazio e raffreddamento dei data center.
Tradizionalmente, gli FPGA richiedono una profonda competenza di dominio per la programmazione. Per semplificare il flusso di sviluppo e consentire un deployment rapido nel data center, Intel offre una piattaforma di accelerazione che include schede di accelerazione programmabili basate su FPGA PCI Express* (PCIe*) (Intel FPGA PAC) e Intel® Acceleration Stack per CPU Intel Xeon® con FPGA. Queste piattaforme Intel sono qualificate, convalidate e implementate tramite Dell EMC. Insieme ai partner dell'ecosistema come Falcon Computing, Intel Acceleration Platform offre una soluzione affidabile e pronta all'uso con hardware trasparente sotto il cofano.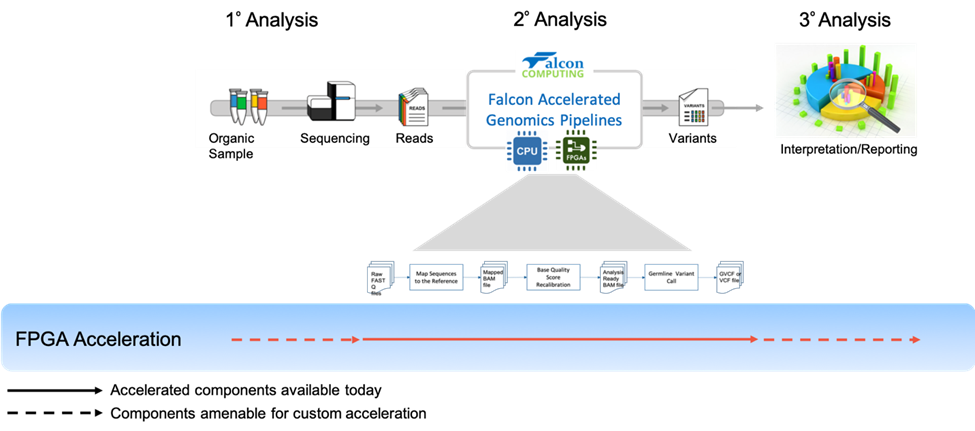
Figura 1: Maggiore precisione e velocità su pipeline GATK standard
Falcon Solution Details:
Il Genome Analysis Toolkit (GATK) è il gold standard per l'elaborazione dei dati genomici accettato dalla comunità genomica (2). Il suo Best Practice Workflow (BPW) è noto per la sua lentezza nel calcolo per generare risultati per campioni di grandi dimensioni come Whole-Genome (WGS). Per affrontare questo problema, Falcon Computing Solutions ha sviluppato un pacchetto software flessibile di strumenti che segue il BPW e può essere implementato in più piattaforme e architetture. È veloce di diversi ordini di grandezza rispetto alle pipeline GATK basate su CPU.
FAGP fornisce una soluzione end-to-end per analizzare i dati genomici a costi contenuti utilizzando la pipeline GATK con prestazioni, precisione e riproducibilità elevate. La soluzione offre una velocità fino a 15 volte superiore con la stessa precisione di GATK (3). Ciò significa che un'analisi che in genere richiede dalle 50 alle 60 ore può essere condotta in meno di 4 ore (3). FAGP fornisce livelli eccezionali di accelerazione e precisione con FPGA Intel Arria 10 affidabili e a prestazioni elevate e processori Intel® Xeon®.
FAGP segue GATK BPW. Implementa l'accelerazione in molti componenti delle pipeline dall'allineamento (BWA) alla chiamata delle varianti (HaplotypeCaller) (4). Oltre al BWA accelerato, include anche una versione accelerata dell'allineatore Minimap2 che fa parte della Alternate Genomic Pipeline di Falcon (5). La pipeline alternativa offre una soluzione ancora più rapida. Può completare il sequenziamento dell'intero genoma 50x entro 3 ore. Entrambi gli allineatori hanno la funzione di generare duplicati contrassegnati e letture ordinate senza la necessità di utilizzare strumenti aggiuntivi.
FAGP raggiunge prestazioni/throughput elevati accelerando il calcolo intensivo nella pipeline GATK utilizzando piattaforme PAC Intel FPGA. Ciò è diverso dalle soluzioni scale-out che raggiungono un throughput elevato aggiungendo più risorse della CPU. Tali soluzioni scale-out hanno una capacità limitata di ridurre i costi o la latenza per campione.
Un altro vantaggio della soluzione Falcon è che si tratta di una pipeline aperta come GATK. Gli utenti possono controllare i singoli passaggi nelle pipeline. I dati intermedi vengono salvati e sono accessibili.
Tabella 1: Vantaggi della pipeline di genomica accelerata Falcon
| Vantaggi di Falcon Accelerated Genomics Pipeline (FAGP) | |
|---|---|
| Vero GATK | Supporto per più versioni di GATK, inclusa la 4.0 |
| Su scala industriale | Esegui cinque genomi interi o 24 esomi interi in un giorno. |
| Variante alternativa | < Tempo di risposta in loco di 3 ore per WGS (50X) |
| Velocità | Esegui la pipeline delle best practice GATK fino a >15 volte più velocemente. |
| Utilizzo ottimale | Non c'è bisogno di riscrivere algoritmi funzionanti. |
Configurazione hardware Dell
Tabella 2. Dell EMC PowerEdge R740xd come banco di prova
| Dell EMC PowerEdge R740xd | |
|---|---|
| Processore | 2x Intel(R) Xeon(R) Gold 6148 CPU @ 2.40GHz |
| Memoria | 384GB @ 32x 16GB RDIMM, 2666MT/s, Dual Rank |
| Storage | 4x 1.2TB 10K RPM SAS 12Gbps 512n 2.5in Hot-plug Hard Drive in RAID 0 2x INTEL SSDPEDMD020T4 DC P3700 1.8T in software RAID 0 |
| FPGA | Intel Programmable Acceleration Card with Intel Arria® 10 GX FPGA (Intel Acceleration Stack 1.1) |
| Profilo di sistema | Performance |
| Versione del BIOS | 2.1.3 |
| Hyper-threading | Enabled |
| Sistema operativo | Red Hat Enterprise Linux Server release 7.4 (Maipo) (3.10.0-693.el7.x86_64) |
Valutazione delle prestazioni
Nei nostri test di benchmark, abbiamo utilizzato dati di sequenziamento dell'intero genoma umano a una profondità di copertura 10x, 30x e 50x.
Tabella 3: Dati di sequenziamento dell'intero genoma testati
| Esegui l'accesso | Profondità della copertura | Collegamento dati |
|---|---|---|
| ERR091571 | 10 volte | https://www.ebi.ac.uk/ena/data/view/ERR091571 |
| SRR3124837 | 30 volte | https://www.ebi.ac.uk/ena/data/view/SRR3124837 |
| ERR194161 | 50 volte | https://www.ebi.ac.uk/ena/data/view/ERR194161 |
Risultati:
La tabella 4 riepiloga il tempo impiegato per completare la pipeline delle best practice GATK 4.0 in tre cicli di test utilizzando FAGP e il PAC FPGA Intel ospitato nel server DELL EMC PowerEdge R740xd.
Tabella 4: Runtime totali da Best Practice Pipeline versione 2.1.1
| Campione | Profondità della copertura | Prova 1 | Tempo di esecuzione (minuti) Test 2 |
Prova 3 |
|---|---|---|---|---|
ERR091571 |
10x |
75.63 |
76.67 |
76.38 |
SRR3124837 |
30x |
160.00 |
162.77 |
161.38 |
ERR194161 |
50x |
242.97 |
250.65 |
247.18 |
La tabella 5 riepiloga il tempo (in minuti) impiegato per completare la pipeline alternativa: Falcon Germline in tre cicli di test utilizzando FAGP e il PAC FPGA Intel alloggiato nel server DELL EMC PowerEdge R740xd.
Tabella 5: Runtime totali dalla pipeline di chiamata di varianti alternative
| Campione | Profondità della copertura | Prova 1 | Tempo di esecuzione (minuti) Test 2 |
Prova 3 |
|---|---|---|---|---|
ERR091571 |
10x |
62.70 |
58.21 |
59.80 |
SRR3124837 |
30x |
130.38 |
129.90 |
129.95 |
ERR194161 |
50x |
171.52 |
171.87 |
171.37 |
Riepilogo della soluzione genomica Falcon
Falcon Accelerated Genomics Pipeline offre un'elevata produttività, vantaggi a basso costo/campione/giorno. Insieme alla scheda di accelerazione programmabile Intel FPGA e al server DELL certificato, FAGP fornisce una soluzione completa che può essere adottata per le applicazioni di sequenziamento genomico".
In TCGB, forniamo servizi di sequenziamento del genoma ai nostri clienti a livello nazionale. La Falcon Accelerated Genomics Pipeline* ci ha permesso di ridurre i tempi di consegna da giorni a poche ore, mantenendo al contempo l'accuratezza delle pipeline GATK standard del settore".
— Dott. Xinmin Li, Direttore del Centro tecnologico per la genomica e la bioinformatica (TCGB) UCLA
Risorse
1. Il sequenziamento del genoma crea così tanti dati che non sappiamo cosa farne. [Online] https://www.washingtonpost.com/news/speaking-of-science/wp/2015/07/07/sequencing-the-genome-creates-so-much-data-we-don't-know-what-to-do-with-it.
2. GATK [In linea] https://software.broadinstitute.org/gatk/
3. Genomica accelerata [in linea] http://www.falconcomputing.com/falcon-accelerated-genomics-pipeline
4. BWA. [In linea]
http://bio-bwa.sourceforge.net/bwa.shtml5. Minimap2. [In linea] https://github.com/lh3/minimap2